[ English | Japanese ]
 遺伝子や蛋白質のシークエンス情報がデータベースに蓄積されるに従い、2次元電気泳動や質量分析といったプロテオミクス手法を用い、問題となる全蛋白質(プロテオーム)を系統的・網羅的に解析することが可能となってきました。最近、こうしたプロテオミクスの手法を応用した蛋白質性アレルゲン群(アレルゲノーム; Allergenome)の迅速・網羅的な解析を、アレルゲノミクス(Allergenomics)と呼ぶことが提案されました(図)。アレルゲノミクスでは、単にアレルゲン候補蛋白質(患者が持つIgE抗体と特異的に相互作用する蛋白質群)を一斉に検出・同定するだけでなく、アレルギー反応の誘発原因となる動植物等に含まれる蛋白質抗原の、状況や環境の変化に伴う質的・量的変動についても追跡することが可能です[1]。
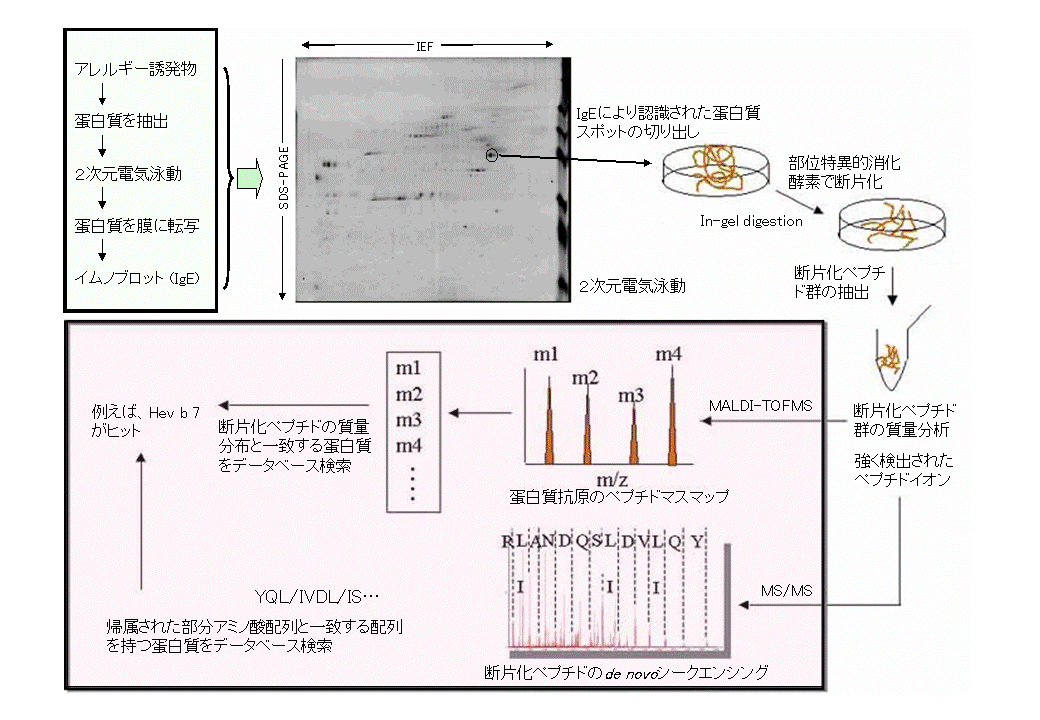
遺伝子や蛋白質のシークエンス情報がデータベースに蓄積されるに従い、2次元電気泳動や質量分析といったプロテオミクス手法を用い、問題となる全蛋白質(プロテオーム)を系統的・網羅的に解析することが可能となってきました。最近、こうしたプロテオミクスの手法を応用した蛋白質性アレルゲン群(アレルゲノーム; Allergenome)の迅速・網羅的な解析を、アレルゲノミクス(Allergenomics)と呼ぶことが提案されました(図)。アレルゲノミクスでは、単にアレルゲン候補蛋白質(患者が持つIgE抗体と特異的に相互作用する蛋白質群)を一斉に検出・同定するだけでなく、アレルギー反応の誘発原因となる動植物等に含まれる蛋白質抗原の、状況や環境の変化に伴う質的・量的変動についても追跡することが可能です[1]。典型的なアレルゲノミクスは、即時型アレルギー反応の誘起物から蛋白質を抽出し、2次元電気泳動法により分離することから始まります(図)。まず1次元目として、固定化pH勾配ゲル(IPGゲル)を用いた等電点電気泳動(isoelectric focusing; IEF)を行い、蛋白質をその等電点(pI)に基づき分離します。さらに、続く2次元目としてSDS-PAGEを行い、蛋白質をその分子量に基づいて分離します。こうして2次元に展開した蛋白質をメンブレンに転写後、適当なアレルギー患者のプール血清と反応させ、IgE抗体と特異的に相互作用した蛋白質抗原を検出します。次に、同様に2次元に展開後、色素染色や蛍光染色したゲルから、IgE結合性蛋白質に相当するスポットを切り出します。そして、ゲル片に含まれる蛋白質を、トリプシンなどの部位特異的蛋白質分解酵素で処理して断片化します(ゲル内消化)。このようにして得られた断片化ペプチド群をゲルから抽出後、MALDI-TOFMSなどで分析し、蛋白質の指紋とも言える部位特異的断片化ペプチドのマススペクトルパターン(ペプチドマスマップ)を得ます。さらに、強く検出された幾つかの断片化ペプチドイオンのMS/MSスペクトルを測定します。最後に、こうした質量分析の結果を基にペプチドマスフィンガープリント (PMF)やペプチドシークエンスタグ (PST)と呼ばれる手法を用いてデータベース検索を行い、元のIgE結合性蛋白質群(アレルゲノーム; allergenome)を同定します。
現在のところアレルゲノミクスには、遺伝子や蛋白質のシークエンス情報がデータベースに多く登録されていない種では通常の検索によるアレルゲン蛋白の網羅的な同定が困難である、という問題点が残っています。しかし、データベース検索で同定できなかった蛋白質についても、断片化ペプチドのMS/MSスペクトルから部分アミノ酸配列に関する情報を得ることは可能であり(de novo シークエンシング)、この部分アミノ酸配列に相同なシークエンスを有する他種由来の蛋白質をデータベース検索することができます。また、部分アミノ酸配列情報を元にオリゴマープローブを作成し、問題となるアレルゲン候補蛋白をコードするcDNAをクローニングして、その全配列を明らかにするといった展開も期待されます。同定されたアレルゲン候補蛋白質が実際にアレルギー反応を誘発するものであるかどうかは、ヒスタミン遊離テストや患者の同意を得て行う皮膚テストなどで確認する必要があります。
国立医薬品食品衛生研究所療品部では、各種の天然ゴム製品や植物性食品により誘発される即時型アレルギー反応であるラテックスアレルギーをモデルケースとして取り上げ、アレルゲン候補蛋白質の迅速・網羅的な解析にアレルゲノミクスがどの程度有効であるか、また解決すべき課題は何かという点について研究を進めています[2]。
[2] 矢上 健, 蓜島由二, 土屋利江, 冨高晶子, 加野尚生, 松永佳世子: プロテオミクスの手法を用いたラテックスアレルゲンの解析, 日本ラテックスアレルギー研究会会誌, Vol.7, No.1, 38-43 (2003); Yagami,T., Haishima, Y., Tsuchiya, T., Tomitaka-Yagami, A., Kano, H. and Matsunaga, K.: Proteomic analysis of putative latex allergens., Int. Arch. Allergy Immunol., 135, 3-11 (2004); 矢上 健, 矢上晶子, 松永佳世子: ラテックス蛋白のアレルゲノミクス, アレルギーの臨床, 25, 306-311 (2005).